 Erkrankungen des Blutkreislaufs gehören zu den am weitesten verbreiteten Krankheiten in Industrienationen. So starben alleine in Deutschland im Jahr 2001 insgesamt 290.000 Personen an Erkrankungen des Herz-Kreislauf-Systems. Zuverlässige Diagnosesysteme und eine Verbesserung bestehender Verfahren sind daher von großer Bedeutung. Eine zentrale Aufgabe stellt hierbei eine geeignete Visualisierung des segmentierten Blutflußbaums beziehungsweise einzelner, erkrankter Abschnitte der Blutgefäße dar.
Erkrankungen des Blutkreislaufs gehören zu den am weitesten verbreiteten Krankheiten in Industrienationen. So starben alleine in Deutschland im Jahr 2001 insgesamt 290.000 Personen an Erkrankungen des Herz-Kreislauf-Systems. Zuverlässige Diagnosesysteme und eine Verbesserung bestehender Verfahren sind daher von großer Bedeutung. Eine zentrale Aufgabe stellt hierbei eine geeignete Visualisierung des segmentierten Blutflußbaums beziehungsweise einzelner, erkrankter Abschnitte der Blutgefäße dar.
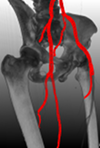
Diese Diplom-/Masterarbeit ist Teil eines Projekts zur vollautomatischen Segmentierung und Interpretation des arteriellen Gefäßbaums. Hierzu werden Verfahren entwickelt um unter Verwendung von anatomischen Hintergrundwissen, das in einem Referenzmodell abgelegt ist, den Gefäßbaum zu segmentieren. Anschließend soll die Diagnose durch geeignete Interpretation der Segmentierung optimal unterstützt werden. Die Grundlage des Systems sind dabei CT-Bilder, in denen Gefäße durch Kontrastmittel von umgebendem Gewebe hervorgehoben dargestellt werden.
Der Aufgabenbereich dieser Diplom-/Masterarbeit umfasst folgende Punkte:
- Effiziente Erzeugung eines Oberflächennetzes aus einem Graphen bestehend aus Gefäßmittelpunkten und zugehörigen Konturen
- 3D Visualisierung des Oberflächennetzes mit Hervorhebung der Abweichungen zwischen Segmentierung und Referenzmodell
- Visualisierung pathologischer Bereiche, wie Stenosen, Aneurysmen und Kalzifikationen
Voraussetzung für eine erfolgreiche Durchführung der Arbeit sind Kenntnisse der Computergraphik/Visualisierung sowie der Programmierung mit C++, wünschenswert ist außerdem Erfahrung in medizinischer Bildverarbeitung
Literatur: T. Beck, C. Biermann, D. Fritz et al.: “Robust model-based centerline extraction of vessels in CTA data”, Proc. SPIE, Vol. 7259, 2009
S. Grosskopf, C. Biermann, K. Deng et al.: “Accurate, fast, and robust vessel contour segmentation of CTA using and adaptive self-learning edge model”, Proc. SPIE, Vol. 7259, 2009
